Tendencias globales en la aplicación de imágenes de fluorescencia en enfermedades pancreáticas: un análisis bibliométrico y de gráficos de conocimiento
tiene visualizaciones que me pueden servir
Tendencias globales en la aplicación de imágenes de fluorescencia en enfermedades pancreáticas: un análisis bibliométrico y de gráficos de conocimiento
tiene visualizaciones que me pueden servir
Terapia antiviral para el virus COVID-19: revisión narrativa y análisis bibliométrico
2019 to 2022
3 años
DOI: 10.1016/S2007-5057(13)72694-2
Cienciometría para ciencias médicas: definiciones, aplicaciones y perspectivas
Layla Michán, Israel Muñoz-Velasco
COVID-19 knowledge deconstruction and retrieval: an intelligent bibliometric solution
Deconstrucción y recuperación del conocimiento COVID-19: una solución bibliométrica inteligente
Drug repurposing for COVID-19 via knowledge graph completion
Reutilización de medicamentos para COVID-19 mediante la finalización del gráfico de conocimiento
Trends in the application of deep learning networks in medical image analysis: Evolution between 2012 and 2020
Tendencias en la aplicación de redes de aprendizaje profundo en el análisis de imágenes médicas: Evolución entre 2012 y 2020
Papeles prometedores de Zingiber officinale roscoe, Curcuma longa L. y Momordica charantia L. como moduladores de la inmunidad contra el COVID-19: un análisis bibliométrico
Mapping the Emergence of Synthetic Biology
Mapeo del surgimiento de la biología sintética
ver si esta en la consulta
TipoDeMetodo: network analysis
BaseDatos: PubMed
CantidadRegistros: 15387
Aplicación: R script
Gephi 0.9.1 and R igraph 0.7.0 package
Aplicación
R script
Aplicación
query = “COVID-19” OR “SARS-CoV-2”
consulta
25 May 2020
Fecha del estudio
Entrez Programming Utilities (E-utilities)
API
PubMed
15,387 articles
cantidad de registros
Citation and co-citation network analysis
COVID-19 PMC citation network is a directed network with 6,650 articles (nodes) and 25,095 citations (edges). Each circle represent an article, each arch represents a citation, and diameter of circle represents number of citation in citation network. (b) COVID-19 PMC co-citation network is an undirected network. The main article cluster (connected component) consist of 2,811 nodes and 78,844 edges. Graph is filtered to represent 19 major articles in PMC co-citation network (co-citation degree ≥ 600). Each circle represent an article, each arch represents a co-citation relation, thickness of edges represents co-citation frequencies
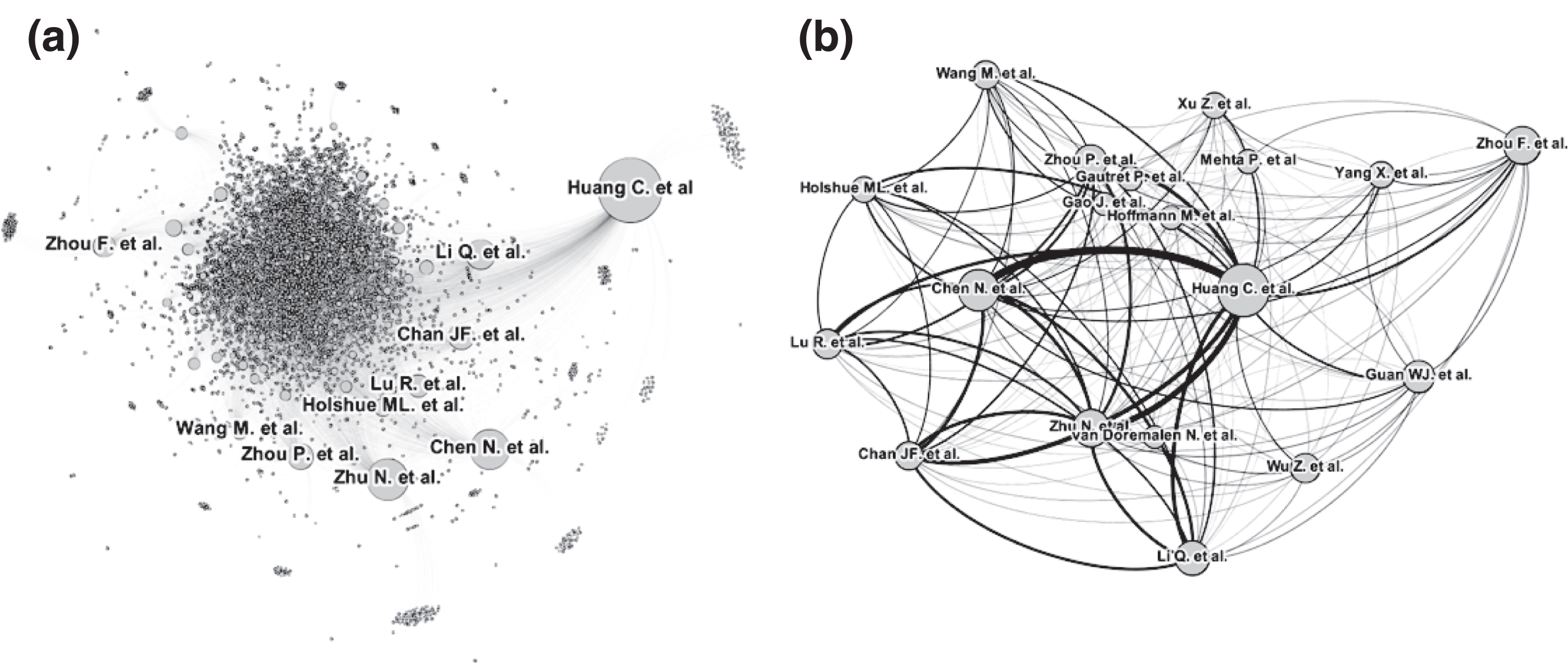
Grafico
2020 a 2021 y 2022 a 2023
Fecha del estudio
Algunas leyes de la bibliometría
Ley de Lotka, (1926) sobre la distribución de la productividad de los autores.
Ley de Zipf (1935) explica cómo se comporta un corpus de lenguaje natural.
Ley de concentración y dispersión de temas en fuentes de información (Bradford 1934)
Multiparametric characterization of scientometric performance profiles assisted by neural networks: a study of Mexican higher education institutions
DOI: 10.1007/s11192-016-2166-0
Caracterización multiparamétrica de perfiles de desempeño cienciométrico asistidos por redes neuronales: un estudio en instituciones de educación superior mexicanas
Elio Atenógenes Villaseñor, Ricardo Arencibia-Jorge & Humberto Carrillo-Calvet
Network Science
Albert-László Barabási
Un libro de texto sobre ciencia de redes, está disponible gratuitamente bajo la licencia Creative Commons.
Salud Pública de México acepta el sometimiento de trabajos previamente publicados como preprints, siempre y cuando los autores lo informen oportunamente, hayan mantenido los derechos patrimoniales de la obra y, en caso de que el manuscrito sea aceptado para publicación, se comprometan a actualizar la información del preprint con un enlace a la versión final publicada en SPM.
Preprints
Patternize: un paquete R para cuantificar la variación del patrón de color.
1900 and 2019
Variable:
fecha de inicio y fecha de fin
895 manuscripts across the 5 journals.
Variable:
numero de registros
articles published in 5 high-performance rheumatology-focused journals in 2019.
Variable:
fuente
Descriptive statistics
Variable:
estadísticas
bibliometric and scientometric analysis
Variable:
metría
scientific research
Variable:
objeto de estudio
VOSviewer
Variable:
software
2007 and 2018
Variable:
fecha de inicio y fecha de fin
Elsevier Scopus
Variable:
fuente
Archives of Microbiology
Variable:
revista
18 Citations
Variable:
numero de citaciones
Joyce Cristina Gonçalvez Roth ORCID: orcid.org/0000-0001-6842-66631, Michele Hoeltz ORCID: orcid.org/0000-0002-6156-26001 & Lisianne Brittes Benitez
Variable:
autores
11 January 2020
Variable:
fecha de publicación
1987 to 2005
Variable:
fecha de inicio y fecha de fin
Conservation BiologyVolume 20, Issue 3 p. 652-657
Variable:
revista
AUTUMN-LYNN HARRISON
Variable:
autores
01 June 2006
Variable:
fecha de publicación
Citations: 29
Variable:
numero de citaciones
This is an Open Access
Variable:
acceso
20 Dec 2021
Variable:
fecha de publicación
VOSviewer (version 1.6.17, 2021, Leiden University, Leiden, The Netherlands)
Variable:
Software
Title: ("Blueberry" or "Blueberries" or "Vaccinium corymbosum L")
Variable:
Consulta
article and review type papers of blueberry
Variable:
tema de estudio
bibliometric analysis; blueberry (Vaccinium corymbosum L.); VOSviewer; Web of Science
Variable:
palabras clave
3,872
Variable:
registros analizados
Web of Science
Variable:
fuente
Alcohol drinkingOral healthOral leukoplakiaOral squamous cell carcinomaTobacco smoking
Variable:
palabras clave
Scopus
Variable:
fuente
2023
Variable:
fecha de publicación
Journal of Dental Sciences
Variable:
revista
Hao Liu a †, Zhonglin Yu b c †, Ziyun Xu c, Tingzhong Liu a, Wei Liu
Variable:
autores
open access
Variable:
acceso
Neotropical Ichthyology
Variable:
revista
This is an open-access article
Variable:
acceso
22 May 2023
Variable:
fecha de publicación
Brazil; Inventory; Scientometric review; Species density; Tropical estuary
Variable:
Palabras clave
PublishingOn-lineBotanyNomenclatureTaxonomy
Variable:
palabras clave
25 May 2017
variable:
fecha de publicación
2009 and 2014
variable:
fecha de inicio y fecha de fin del estudio
International Plant Names Index (IPNI)
Variable:
fuente
6 Citations
Variable:
Número de citaciones
BMC Evolutionary Biology
Variable:
revista
Nicky Nicolson1,3, Katherine Challis2, Allan Tucker3 & …Sandra Knapp
Variable:
autores
2010 to 2020
Variable:
fecha de inicio y fecha de fin
“Web of Science Core Collection” (WoSCC)
Variable:
fuente
Li, Feifei MDa; Wang, Shuqi PhDa; Yao, Youlong MDb; Sun, Xueming MDc; Wang, Xiaoyan MDc; Wang, Ning MDa; You, Yulin MDa; Zhang, Yanli PhD
Variable:
autores
Muy bien. * Procura que todos tus trabajos tengan tu nombre.
faltan las referencias
Macho (Fig. 13 y 14): envergadura alar 35-36 mm. Anverso del ala anterior de color café oscuro con una línea vertical gris claro en el área postmedial que atraviesa toda el ala sin entrar en la celda discal. Anverso de las alas posteriores de color negro desde el margen distal hasta el área medial sin alcanzar la celda discal, con serie de 18 puntos circulares y semicirculares de color gris con tonalidad azul magenta claro, ordenados en forma concéntrica y un par de ocelos negros con pupila blanca ubicados en el margen alar entre las venas R3-M1 y M1-M2 ( en P. lena brasiliensis C. Felder & R.Felder,1862, las manchas son de color azul oscuro con reflejo azul iridiscente). Reverso del ala anterior de color café oscuro en el área postmedial y marginal, con 4 puntos submarginales de color blanco, área postmedial delimitada por una banda delgada de color gris, con borde café oscuro, en forma vertical que atraviesa toda el ala, área medial, postmedial y basal de color gris con 3 puntos blancos en la celda discal (en P. lena brasiliensis el ala es de color café oscuro, con la banda de color gris mas delgada y tenue). Reverso del ala posterior de color café oscuro en el área postmedial y marginal, con dos ocelos de color negro con borde amarillo y pupila blanca. El área media presenta 3 manchas de color crema. El área postmedial se encuentra dividida del área medial por una banda gris con borde café oscuro, que atraviesa toda el ala posterior. Área submedial de color gris con una línea oblicua delgada de color café oscuro y 4 puntos café en el área basal. Hembra (Fig. 15): envergadura alar 37-39 mm. Anverso y reverso de las alas anteriores con patrón de coloración igual que en el macho. Anverso de las alas posteriores de color negro en casi toda el ala y con puntos grises más pequeños que en el macho, con tonalidad azul magenta, distribuidos en la misma posición que en el macho.
Pierella lena salma
Lepidoptera
DiagnosisBIOcolores
https://www.wikidata.org/wiki/Q21359095
Pierella lena brasiliensis
envergadura alar 36-37 mm. Anverso del ala anterior completamente transparente. Anverso del ala posterior con una mancha amarilla que se extiende desde el margen anal hasta la vena M2 y la celda discal sin entrar en ella. Dos ocelos negros con pupila blanca en el margen alar entre las venas M1-M2 y Cu1-Cu2, siendo las primeras de mayor tamaño. Presenta una línea submarginal café claro interrumpida y muy tenue que se extiende desde la vena R3 hasta la vena 2A. (En H. piera negra Felder & Felder, 1862 la línea café es continua y más fuerte y la mancha amarilla más tenue y menos extensa que en H. piera sanguinolenta (Fig. 3). Hembra (Fig. 2): envergadura alar 39-40 mm. Anverso del ala anterior completamente transparente. Anverso del ala posterior con una mancha rojo-escarlata intensa en forma de roseta en el torno del ala, delimitada por una banda negra ancha de color negro, muy similar al de la hembra de H. macleannania Bates, 1865 (Fig. 4). Mancha amarilla muy reducida y dos ocelos negros con pupila blanca en el margen alar entre las venas M1-M2 y Cu1-Cu2 de mayor tamaño que en los machos (H. piera negra no presenta mancha roja en el torno del ala posterior y la línea café submarginal es tenue y no negra como en H. piera sanguinolenta. Los ocelos en H. piera negra son mas reducidos en tamaño y la mancha amarilla mas extendida).
Haetera piera sanguinolenta
Lepidoptera
DiagnosisBIOcolores
lepidoptera
Orugas
https://royalsocietypublishing.org/doi/10.1098/rspb.2022.2293
Lista de consultas SPARQL Sobre bases de datos
MinervaMaríaRomeroPérez
Manual de curación usando Open Refine
MinervaMaríaRomeroPérez
Manual de curación usando Quick Statements
MinervaMaríaRomeroPérez
Seminario Bioinformación: El wikiproyecto bases de datos de biociencias
MinervaMaríaRomeroPérez
Proyecto de Wikidata Bases de datos de biociencias
LaylaMichánAguirre
MinervaMaríaRomeroPérez
Formulario de wikidata para bases de datos de Biociencias
MinervaMaríaRomeroPérez
Manual Biodatabases Wikidata
MinervaMaríaRomeroPérez
http://colsci.weebly.com/contact.html
Laboratorio:
wd:https://www.wikidata.org/wiki/Q113624269
Investigador:
mencionado en: A Survey of Color Charts for Biological Descriptions
narid:https://www.oxfordjournals.org/nar/database/summary/1095
narCategory:Category: Metabolic and Signaling Pathways
Subcategory: Metabolic pathways
Category: Plant databases
Subcategory: General plant databases
narid:https://www.oxfordjournals.org/nar/database/summary/1112
narCategory:Category: Metabolic and Signaling Pathways
Subcategory: Signalling pathways
Category: Plant databases
Subcategory: Arabidopsis thaliana
narid:https://www.oxfordjournals.org/nar/database/summary/91
https://www.oxfordjournals.org/nar/database/summary/1521
narCategory:Category: Metabolic and Signaling Pathways
Subcategory: Protein-protein interactions
narid:https://www.oxfordjournals.org/nar/database/summary/1046
narCategory:Metabolic and Signaling Pathways
Subcategory: Protein-protein interactions
Category: Plant databases
Subcategory: Arabidopsis thaliana
narid:https://www.oxfordjournals.org/nar/database/summary/985
narCategory: Human and other Vertebrate Genomes
Subcategory: Human genome databases, maps and viewers
Category: Human and other Vertebrate Genomes
Subcategory: Human ORFs
narid:https://www.oxfordjournals.org/nar/database/summary/1055
narCategory:Microarray Data and other Gene Expression Databases
Category: Other Molecular Biology Databases
Subcategory: Drugs and drug design
narid:https://www.oxfordjournals.org/nar/database/summary/1320
narCategory: Microarray Data and other Gene Expression Databases
Category: Organelle databases
Subcategory: Mitochondrial genes and proteins
narid:https://www.oxfordjournals.org/nar/database/summary/1976
https://www.oxfordjournals.org/nar/database/summary/1991
narCategory:Other Molecular Biology Databases
Subcategory: Drugs and drug design
Category: Proteomics Resources
narid:https://www.oxfordjournals.org/nar/database/summary/722
narCategory: Organelle databases
Category: Plant databases
Subcategory: Arabidopsis thaliana
NARid: https://www.oxfordjournals.org/nar/database/summary/1311
NARCategory: Structure Databases
Subcategory: Protein structure
narid:https://www.oxfordjournals.org/nar/database/summary/618
narCategory:Nucleotide Sequence Databases
Subcategory: Transcriptional regulator sites and transcription factors
Category: Plant databases
Subcategory: Arabidopsis thaliana
narid:https://www.oxfordjournals.org/nar/database/summary/1935
narCategory: Protein sequence databases
Subcategory: Databases of individual protein families
narid:https://www.oxfordjournals.org/nar/database/summary/221
narCategory:Protein sequence databases
Subcategory: Protein properties
Category: Proteomics Resources
narid:https://www.oxfordjournals.org/nar/database/summary/452 (Nota: la URL de narid es incorrecta)
narCategory:Protein sequence databases
Subcategory: Protein sequence motifs and active sites
Category: Structure Databases
Subcategory: Protein structure
narid:https://www.oxfordjournals.org/nar/database/summary/873
narCategory:Genomics Databases (non-vertebrate)
Subcategory:Unicellular eukaryotes genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/2116
narCategory:Genomics Databases (non-vertebrate)
Subcategory: Model organisms, comparative genomics
narid:https://www.oxfordjournals.org/nar/database/summary/1149
narCategory:RNA sequence databases, Structure Databases
Subcategory:RNA sequence databases, Nucleic acid structure
narid:https://www.oxfordjournals.org/nar/database/summary/1503
narCategory:Genomics Databases (non-vertebrate)
Subcategory:Viral genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/123
https://www.oxfordjournals.org/nar/database/summary/1795
narCategory:Genomics Databases (non-vertebrate)
Subcategory: Invertebrate genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/1945
narCategory:Genomics Databases (non-vertebrate)
Subcategory: Viral genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/1784
narCategory:Metabolic and Signaling Pathways
Subcategory: Protein-protein interactions
narid:https://www.oxfordjournals.org/nar/database/summary/1787
narCategory:Proteomics Resources
Subcategory:Proteomics Resources
narid:https://www.oxfordjournals.org/nar/database/summary/900
narCategory: RNA sequence databases
Category: Structure Databases
Subcategory: Nucleic acid structure
narid:https://www.oxfordjournals.org/nar/database/summary/900
narCategory: RNA sequence databases
Category: Structure Databases
Subcategory: Nucleic acid structure
narid:https://www.oxfordjournals.org/nar/database/summary/47
narid:https://www.oxfordjournals.org/nar/database/summary/705
narCategory:RNA sequence databases
narid:https://www.oxfordjournals.org/nar/database/summary/424
narCategory: Genomics Databases (non-vertebrate)
Subcategory: Prokaryotic genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/1734
narCategory:Other Molecular Biology Databases
narid:https://www.oxfordjournals.org/nar/database/summary/1581
narCategory:Category: Microarray Data and other Gene Expression Databases
narid:https://www.oxfordjournals.org/nar/database/summary/1507
(Nota: la URL de narid es incorrecta)
narCategory:Genomics Databases (non-vertebrate)
Subcategory: Prokaryotic genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/1924
(Nota: la URL de narid es incorrecta)
narCategory:Nucleotide Sequence Databases
Subcategory: Coding and non-coding DNA
narid:https://www.oxfordjournals.org/nar/database/summary/1153
narCategory:Genomics Databases (non-vertebrate)
Subcategory:Prokaryotic genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/1647
narCategory:Microarray Data and other Gene Expression Databases
narid:https://www.oxfordjournals.org/nar/database/summary/1829
narCategory:Cell biology
Subcategory:Cell biology
narid:https://www.oxfordjournals.org/nar/database/summary/991 narCategory:Metabolic and Signaling Pathways
Subcategory: Metabolic pathways
narid:https://www.oxfordjournals.org/nar/database/summary/92
narCategory: Metabolic and Signaling Pathways
Subcategory:Enzymes and enzyme nomenclature
narid:https://www.oxfordjournals.org/nar/database/summary/641
(Nota: la URL en NAR esta mal pues esta es una nueva versión la pagina anterior)
narCategory:Genomics Databases (non-vertebrate)
Subcategory:General genomics databases
narid:https://www.oxfordjournals.org/nar/database/summary/257
narCategory:Structure Databases
Subcategory:Protein structure
narid:https://www.oxfordjournals.org/nar/database/summary/1718
narCategory:Human Genes and Diseases
Subcategory:General human genetics databases
narid:https://www.oxfordjournals.org/nar/database/summary/1189
narCategory:Metabolic and Signaling Pathways
Subcategory:Enzymes and enzyme nomenclature
narid:https://www.oxfordjournals.org/nar/database/summary/1660
narCategory:Plant databases
Subcategory:Rice
narid:https://www.oxfordjournals.org/nar/database/summary/931
Category: Nucleotide Sequence Databases
Subcategory: Coding and non-coding DNA
Category: Genomics Databases (non-vertebrate)
Subcategory: Fungal genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/298
narCategory:Other Molecular Biology Databases
Subcategory:Other Molecular Biology Databases
narid:https://www.oxfordjournals.org/nar/database/summary/242
narCategory:Structure Databases
Subcategory:Nucleic acid structure
narid:https://www.oxfordjournals.org/nar/database/summary/2008
narCategory:Genomics Databases (non-vertebrate)
Subcategory:TGeneral genomics databases
narid:https://www.oxfordjournals.org/nar/database/summary/697
(Nota: la URL de narid es incorrecta)
narid:https://www.oxfordjournals.org/nar/database/summary/917
(Nota: la URL de narid es incorrecta)
narCategory:Genomics Databases (non-vertebrate)
Subcategory:General genomics databases
narid:https://www.oxfordjournals.org/nar/database/summary/127
https://www.oxfordjournals.org/nar/database/summary/1151
narCategory:Human and other Vertebrate Genomes
Subcategory:Model organisms, comparative genomics
narid:https://www.oxfordjournals.org/nar/database/summary/1631
narCategory:Metabolic and Signaling Pathways
Subcategory:Metabolic pathways
narid:https://www.oxfordjournals.org/nar/database/summary/645
narCategory:Genomics Databases (non-vertebrate)
Subcategory:Fungal genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/355
narCategory:Plant databases
Subcategory:General plant databases
narid:https://www.oxfordjournals.org/nar/database/summary/50
narCategory:RNA sequence databases
Subcategory:RNA sequence databases
narid:https://www.oxfordjournals.org/nar/database/summary/860 narCategory:Plant databases
Subcategory:Rice
narid:https://www.oxfordjournals.org/nar/database/summary/490
narCategory:Genomics Databases (non-vertebrate), RNA sequence databases
Subcategory:General genomics databases, RNA sequence databases
narid:https://www.oxfordjournals.org/nar/database/summary/526
narCategory:Plant databases
Subcategory:Other plants
narid:https://www.oxfordjournals.org/nar/database/summary/1511
narCategory:Genomics Databases (non-vertebrate)
Subcategory:Fungal genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/1997
narCategory:Genomics Databases (non-vertebrate)
Subcategory:Prokaryotic genome databases
http://cpdb.molgen.mpg.de/
narid:https://www.oxfordjournals.org/nar/database/summary/1227
narCategory:Metabolic and Signaling Pathways
Subcategory:Protein-protein interactions
narid:https://www.oxfordjournals.org/nar/database/summary/1431
narCategory:Metabolic and Signaling Pathways
Subcategory:Protein-protein interactions
narid:https://www.oxfordjournals.org/nar/database/summary/570
narCategory:Genomics Databases (non-vertebrate)
Subcategory:Invertebrate genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/1700
narCategory:Genomics Databases (non-vertebrate)
Subcategory:Prokaryotic genome databases
narid:https://www.oxfordjournals.org/nar/database/summary/1995
narCategory:Genomics Databases (non-vertebrate)
Subcategory:General genomics databases
narid:https://www.oxfordjournals.org/nar/database/summary/957
narCategory:Genomics Databases (non-vertebrate)
Subcategory:General genomics databases
narid:https://www.oxfordjournals.org/nar/database/summary/620
narCategory:Plant databases
Subcategory:Other plants
narid:https://www.oxfordjournals.org/nar/database/summary/646
narCategory:Structure Databases
Subcategory:Small molecules
narid:https://www.oxfordjournals.org/nar/database/summary/1183
narCategory:Genomics Databases (non-vertebrate)
Subcategory:Taxonomy and identification
narid:https://www.oxfordjournals.org/nar/database/summary/1462
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/1339
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/1765
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors
http://www.tractor.lncc.br/
narid:https://www.oxfordjournals.org/nar/database/summary/756
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/904
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/1334
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/1015
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/1582
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/246
narCategory:Nucleotide Sequence Databases, RNA sequence databases
Subcategory:TTranscriptional regulator sites and transcription factors, RNA sequence databases
narid:https://www.oxfordjournals.org/nar/database/summary/577
narCategory:Genomics Databases (non-vertebrate), Nucleotide Sequence Databases
Subcategory:Fungal genome databases, Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/1461
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/1128
narCategory:Genomics Databases (non-vertebrate), Nucleotide Sequence Databases
Subcategory:Invertebrate genome databases, Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/563
narCategory:Nucleotide Sequence Databases, Plant databases Subcategory:TTranscriptional regulator sites and transcription factors, General plant databases
narid:https://www.oxfordjournals.org/nar/database/summary/1107
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors
narid:https://www.oxfordjournals.org/nar/database/summary/185
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors wd:https://www.wikidata.org/wiki/Q111431789
narid:https://www.oxfordjournals.org/nar/database/summary/1678
narCategory:Nucleotide Sequence Databases
Subcategory:Transcriptional regulator sites and transcription factors